데이콘 LG 농업 환경 변화에 따른 작물 병해 진단 AI 경진대회 분석 코드

📌 코드 공유
데이콘에 분석 코드를 공유했던 링크를 첨부합니다.
https://dacon.io/codeshare/4405
클래스별 이미지 및 환경변수 plot 코드 공유합니다
농업 환경 변화에 따른 작물 병해 진단 AI 경진대회
dacon.io
📌 대회 설명
1. 개요 : "작물 환경 데이터"와 "작물 병해 이미지"를 이용해 "작물의 종류", "병해의 종류", "병해의 진행 정도"를 진단하는 AI 모델 개발
2. 평가 기준 : Macro F1
3. 대회 기간 : 2022.01.12 ~ 2022.02.04
4. 대회 링크 : https://dacon.io/competitions/official/235870/overview/description
📌 목차
1. 데이터의 클래스 종류별 개수 시각화
2. 환경변수의 seq length 시각화 및 클래스별 환경변수 seq length 통계량 확인
2-1. 환경변수의 seq length 시각화
2-2. 클래스별 환경변수의 seq length 통계량 확인
3. 클래스별 이미지 시각화
4. 클래스별 환경변수 시각화
📌 데이터의 클래스 종류별 개수 시각화
✔ train 데이터의 label별 개수
label_description = {
'1_00_0' : '딸기_정상',
'2_00_0' : '토마토_정상',
'2_a5_2' : '토마토_흰가루병_중기',
'3_00_0' : '파프리카_정상',
'3_a9_1' : '파프리카_흰가루병_초기',
'3_a9_2' : '파프리카_흰가루병_중기',
'3_a9_3' : '파프리카_흰가루병_말기',
'3_b3_1' : '파프리카_칼슘결핍_초기',
'3_b6_1' : '파프리카_다량원소결핍(N)_초기',
'3_b7_1' : '파프리카_다량원소결핍(P)_초기',
'3_b8_1' : '파프리카_다량원소결핍(K)_초기',
'4_00_0' : '오이_정상',
'5_00_0' : '고추_정상',
'5_a7_2' : '고추_탄저병_중기',
'5_b6_1' : '고추_다량원소결핍(N)_초기',
'5_b7_1' : '고추_다량원소결핍(P)_초기',
'5_b8_1' : '고추_다량원소결핍(K)_초기',
'6_00_0' : '시설포도_정상',
'6_a11_1' : '시설포도_탄저병_초기',
'6_a11_2' : '시설포도_탄저병_중기',
'6_a12_1' : '시설포도_노균병_초기',
'6_a12_2' : '시설포도_노균병_중기',
'6_b4_1' : '시설포도_일소피해_초기',
'6_b4_3' : '시설포도_일소피해_말기',
'6_b5_1' : '시설포도_축과병_초기',
}
df = pd.read_csv(os.path.join(args.root, 'train.csv'))
df['desc'] = df['label'].map(label_description)
plt.figure(figsize=(10, 6))
sns.countplot(y='desc', data=df)
train 데이터에 존재하는 총 클래스 개수는 25개입니다. 이 중 파프리카·오이·딸기·시설포도의 정상 데이터는 비교적 많은 편이지만 그외 클래스의 데이터가 현저히 적어 데이터 불균형이 심하다는 사실을 알 수 있습니다.
📌 환경변수의 seq length 시각화 및 클래스별 환경변수 seq length 통계량 확인
✔ 환경변수의 seq length 시각화
env_paths = sorted(glob(os.path.join(args.train_data_path, '*/*.csv')))
env_features = ['내부 온도 1 평균', '내부 온도 1 최고', '내부 온도 1 최저',
'내부 습도 1 평균', '내부 습도 1 최고', '내부 습도 1 최저']
env_len = []
for path in tqdm(env_paths):
env_df = pd.read_csv(path)[env_features]
env_len.append(len(env_df))
sns.displot(env_len)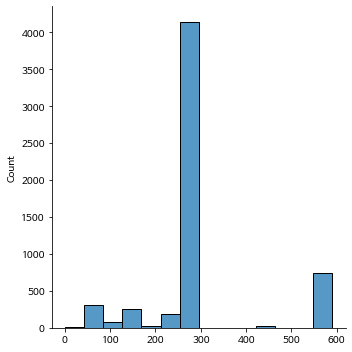
print(f'전체 train 데이터의 환경변수 sequence length max값 : {np.max(env_len)}')
print(f'전체 train 데이터의 환경변수 sequence length min값 : {np.min(env_len)}')
print(f'전체 train 데이터의 환경변수 sequence length mean값 : {np.mean(env_len)}')
print(f'전체 train 데이터의 환경변수 sequence length median값 : {np.median(env_len)}')전체 train 데이터의 환경변수 sequence length max값 : 590
전체 train 데이터의 환경변수 sequence length min값 : 1
전체 train 데이터의 환경변수 sequence length mean값 : 308.32790012138025
전체 train 데이터의 환경변수 sequence length median값 : 294.0
전체 train 데이터의 환경변수의 sequence length 평균은 약 308이며 median 값은 294입니다. 또한 최댓값은 590인 것을 확인할 수 있습니다.
✔ 클래스별 환경변수의 seq length 통계량 확인
len_df = pd.DataFrame({'image' : df['image'].values,
'label' : df['label'].values,
'desc' : df['desc'].values,
'env_len' : env_len})
len_df.groupby(['label', 'desc'])['env_len'].agg(['min', 'max', 'mean', 'median']).reset_index()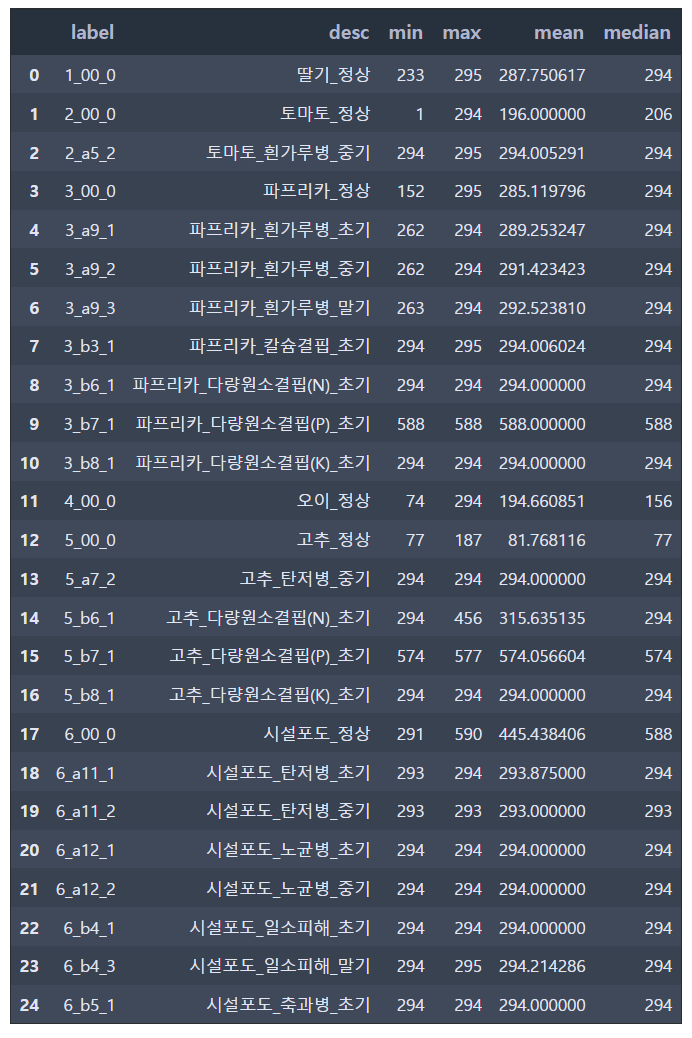
클래스별 환경변수의 sequence length 통계량을 확인해보면 대부분의 클래스의 환경변수 sequence length가 294개 내외이지만 파프리카 다량원소결핍(P) 초기는 평균 588개, 오이 정상은 평균 194.66개, 고추 정상은 평균 81.77개, 고추 다량원소결핍(N) 초기는 평균 315.64개, 고추 다량원소결핍(P) 초기는 평균 574.06개, 시설포도 정상은 평균 445.44개 임을 확인할 수 있습니다.
전체적으로 환경변수의 sequence length을 확인해볼 때, 환경변수 모델링 시 sequence length를 294, 308, 590 에서 선택하는 것이 좋을 것으로 판단됩니다.
📌 클래스별 이미지 시각화
각 label별 임의의 8개의 이미지를 시각화하였습니다.
✔ 이미지 시각화 함수
def visualize_image(disease_code:str, num:int=8):
img_list = list(df.loc[df['label'] == disease_code].sample(num)['image'])
fig, axs = plt.subplots(2, 4, figsize=(12, 7))
fig.suptitle(label_description[disease_code] + f'({disease_code})', fontsize=15)
axs = axs.flatten()
for img_code, ax in zip(img_list, axs):
image = img.imread(os.path.join(args.train_data_path, str(img_code), str(img_code)+'.jpg'))
ax.imshow(image)
ax.set_title(str(img_code))
plt.show()
✔ 딸기 정상 label에 대하여 임의의 8개 이미지 시각화
visualize_image('1_00_0')
딸기는 정상 데이터만 존재합니다.
(...)
visualize_image 함수를 토마토, 파프리카, 오이, 고추, 시설포도에도 적용하여 이미지를 확인 및 분석하였으며 블로그에선 그 결과를 생략합니다. 각각 적용한 결과는 위의 코드 공유 링크를 통해 확인해주세요.
📌 클래스별 환경변수 시각화
1. 각 label별로 임의의 12개 데이터들을 대상으로 6개의 환경변수('내부 온도 1 평균', '내부 온도 1 최고', '내부 온도 1 최저', '내부 습도 1 평균', '내부 습도 1 최고', '내부 습도 1 최저')를 plot 시각화하였습니다. label별 모든 데이터가 아닌 임의의 12개 데이터를 plot화한 것이니만큼 이를 통해 각 label의 특징을 일반화할 수는 없으나 label별 대략적인 경향성을 가시적으로 파악하기에 용이합니다.
2. plot 상의 y축은 온도 또는 습도를 의미하며 x축은 sequence length를 의미합니다. x축의 경우 관측 초기부터 최신으로 정렬하였습니다. (x축에서 0이 관측 초기, x축 sequence length 최댓값이 관측 최신 데이터입니다.)
3. 데이터마다 sequence length가 다른 경우들이 있어서 어떤 데이터들은 그래프상 중간에 잘린 것처럼 보이기도 한다는 점을 참고 바랍니다. 또한 plot 상 직선으로 보이는 데이터는 모든 시계열 상에 동일한 값으로 채워진 데이터임을 참고 바랍니다.
✔ label별 환경변수 시각화 함수
def plot_env_features(disease_code:str, num:int=12):
img_list = list(df.loc[df['label'] == disease_code]['image'].sample(num))
env_features = ['내부 온도 1 평균', '내부 온도 1 최고', '내부 온도 1 최저', '내부 습도 1 평균', '내부 습도 1 최고', '내부 습도 1 최저']
fig, axs = plt.subplots(2, 3, figsize=(16, 7))
fig.suptitle(label_description[disease_code] + f'({disease_code})', fontsize=14)
for img_code in img_list:
_meta = pd.read_csv(os.path.join(args.train_data_path, str(img_code), str(img_code)+'.csv'))[env_features]
_meta = _meta.reindex(index=_meta.index[::-1])
axs[0, 0].plot(_meta['내부 온도 1 평균'])
axs[0, 0].set_title('내부 온도 1 평균')
axs[0, 1].plot(_meta['내부 온도 1 최고'])
axs[0, 1].set_title('내부 온도 1 최고')
axs[0, 2].plot(_meta['내부 온도 1 최저'])
axs[0, 2].set_title('내부 온도 1 최저')
axs[1, 0].plot(_meta['내부 습도 1 평균'])
axs[1, 0].set_title('내부 습도 1 평균')
axs[1, 1].plot(_meta['내부 습도 1 최고'])
axs[1, 1].set_title('내부 습도 1 최고')
axs[1, 2].plot(_meta['내부 습도 1 최저'])
axs[1, 2].set_title('내부 습도 1 최저')
plt.show()
✔ 딸기 정상 label에 대하여 임의의 12개 데이터들을 대상으로 6개의 환경변수 plot 시각화
plot_env_features('1_00_0')
(...)
plot_env_features 함수를 토마토, 파프리카, 오이, 고추, 시설포도에도 적용하여 환경변수를 확인 및 분석하였으며 블로그에선 그 결과를 생략합니다. 각각 적용한 결과는 위의 코드 공유 링크를 통해 확인해주세요.
환경변수를 확인해보니 이미지(육안) 상으로 label의 구분이 어려웠던 데이터라도 환경변수 상의 특징이 존재한다는 것을 확인할 수 있었습니다.
그리고 병해가 있는 작물의 환경변수를 확인해보면 정상 데이터와 비교했을 때 온도와 습도 상의 특징이 있고 어떤 경우엔 이상치들도 존재합니다. 이러한 환경변수의 특징들을 반영해서 모델링을 하면 모델의 학습에 도움이 될 것으로 판단됩니다. 다만 같은 클래스 내에서도 상이한 패턴을 보이는 개체들이 있어서 이런 부분을 잘 처리해주어야 할 것 같습니다.
'공부 > AI' 카테고리의 다른 글
| [Prompt] 프롬프트 엔지니어링 테크닉 기초 정리 (6) | 2023.11.19 |
|---|---|
| [DACON] 유방암의 임파선 전이 예측 AI 경진대회 후기 (7등) (0) | 2023.04.07 |
| [Kaggle] PetFinder.my - Pawpularity Contest 후기 (Top 3%, Silver Medal) (0) | 2023.04.06 |
| [논문리뷰] EfficientNet : Rethinking Model Scaling for Convolutional Neural Networks (2019) (4) | 2023.02.01 |
| [논문리뷰] Deep Residual Learning for Image Recognition (2015) (0) | 2023.01.31 |



